D’une grenouille australienne qui a avalé ses propres œufs aux mammouths laineux, les scientifiques sont de plus en plus près de pouvoir ramener d’entre les morts des espèces disparues depuis longtemps.
Il y a des millions d’années, les thylacines, également connus sous le nom de tigres de Tasmanie, étaient répandus dans toute l’Australie. De la taille d’un coyote américain, ces créatures à rayures ressemblant à des chiens ont disparu du continent il y a environ 2 000 ans.
Ils sont restés en Tasmanie jusque dans les années 1920, où ils ont été abattus par les colonisateurs européens qui les considéraient comme une menace pour le bétail.
« Les colons européens sont arrivés en Australie et ont brutalement éliminé cet animal », explique Andrew Pask, généticien à l’université de Melbourne.
M. Pask dirige une équipe de scientifiques qui, en collaboration avec la société Colossal Biosciences, spécialisée dans la « désextinction », a pour objectif de recréer et de ramener à la vie cette créature semblable à un loup.
Grâce aux récents progrès de la génétique, notamment l’avènement de la technologie d’édition de gènes Crispr-Cas9, le thylacine n’est pas la seule espèce disparue que nous pourrions bientôt revoir. Comment fonctionne la science de la dé-extinction, et quels types de questions éthiques soulève-t-elle ?
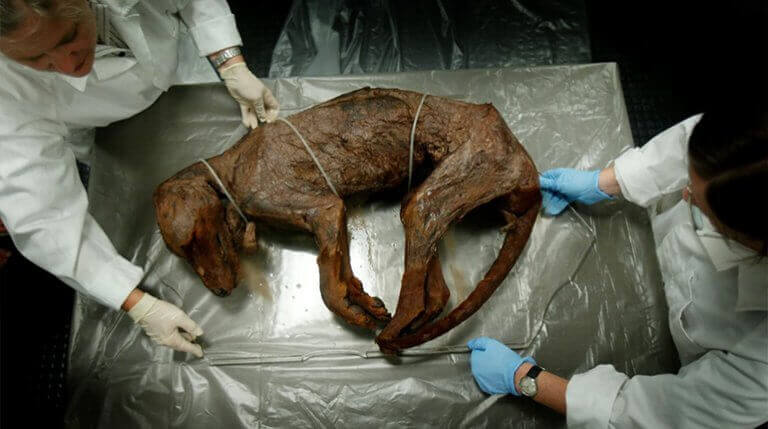
Dans le cas du thylacine, la première étape consiste à séquencer l’ADN de l’animal disparu – le plan génétique contenu dans chaque cellule du corps. Pask l’a fait en 2017.
« Ce qui est génial avec le thylacine, c’est que comme c’était un marsupial si important, tous les grands musées en voulaient un dans leur collection, il y a donc des centaines d’échantillons dans le monde, et certains sont exceptionnellement conservés », explique Pask.
« Notre échantillon était un bébé sorti de la poche de sa mère. Ils ont tiré sur la mère et ont immédiatement plongé le bébé dans l’alcool, qui préserve l’ADN. C’était le spécimen miracle et le Saint-Graal pour nous, en termes de capacité à vraiment construire ce génome. »
Bien qu’il soit en assez bon état, l’ADN n’est pas complètement complet. Avec le temps, l’exposition aux rayons UV et l’action des bactéries décomposent l’ADN en courts fragments. Plus l’échantillon est vieux, plus les fragments sont petits, jusqu’à ce qu’il n’en reste plus assez (il n’y a aucune chance de ramener un dinosaure, pour cette raison).
Les scientifiques sont alors confrontés à la tâche apparemment impossible de comprendre comment les différents morceaux d’ADN s’assemblent – une tâche comparable à celle de compléter un énorme puzzle sans l’image utile sur le devant de la boîte.
Heureusement, un petit marsupial de la taille d’une souris, le dunnart, a pu fournir un modèle. « Nous avons trouvé le plus proche parent vivant du thylacine, le dunnart », explique Pask.
Les dunnarts et les thylacines partagent 95 % de leur ADN, qui est considéré comme hautement conservé, ce qui signifie qu’il n’a pas beaucoup changé au fil du temps.
Personne ne l’a fait à cette échelle auparavant parce que la technologie d’édition de l’ADN n’était pas assez bonne ou assez rapide – Andrew Pask
« Nous avons séquencé le génome du dunnart et comparé son code génétique à celui de notre espèce disparue, puis nous les avons superposés et avons trouvé partout où il était différent », explique Andrew Pask.
Cependant, il ne suffit pas de connaître l’ADN d’un animal pour le faire revivre. La prochaine étape du puzzle consiste à modifier les gènes du dunnart pour qu’ils correspondent à ceux du thylacine. Cela peut être fait avec Crispr-Cas9, la méthode d’édition du génome récompensée par le prix Nobel.
« Nous commençons par des cellules vivantes du dunnart et nous modifions toutes ces modifications, de sorte que nous transformons essentiellement cette cellule de dunnart en une cellule vivante de thylacine contenant des chromosomes de thylacine », explique Pask.
Auparavant, l’édition de gènes n’était pas suffisamment avancée pour permettre de modifier toutes les séquences différentes de l’ADN du thylacine en une seule fois. Des millions de modifications étant nécessaires, on supposait que les chercheurs devraient donner la priorité aux séquences d’ADN les plus importantes, ce qui donnerait un génome animal qui ne serait pas exactement le même que celui de l’animal disparu. M. Pask pense que cela ne sera plus nécessaire.
« Toutes ces technologies sont en place, mais personne ne l’a fait à cette échelle auparavant parce que la technologie d’édition de l’ADN n’était pas assez bonne ou assez rapide. Mais aujourd’hui, le chemin parcouru est si long que nous disposons de cette technologie, et nous avons bénéficié d’investissements importants pour essayer de faire en sorte que cela fonctionne. »
Une fois que les chercheurs disposent d’une cellule de thylacine, ils doivent encore la transformer en un embryon en développement, puis l’implanter dans l’utérus d’un proche parent vivant. Si cela semble facile, ce n’est pas le cas. « Nous avons beaucoup de travail à faire », déclare Pask.
« Nous avons déjà fabriqué des cellules souches de marsupial, ce qui nous a pris environ cinq ans. Nous plaçons maintenant ces cellules souches dans des embryons pour voir si nous pouvons les faire se développer en un animal vivant entier. »
En savoir plus sur les droits des animaux
Il n’y a pas que le thylacine qui pourrait être ramené de cette façon. Des fragments d’ADN de mammouth laineux retrouvés congelés dans la toundra arctique signifient que ce grand mammifère pourrait revenir. La plupart des mammouths laineux se sont éteints il y a environ 10 000 ans.
Les scientifiques de Colossal Laboratories and Bioscience – cofondé par des chercheurs de l’Université de Harvard – utilisent Crispr pour épisser des morceaux d’ADN de mammouth dans le génome de l’éléphant d’Asie, le plus proche parent vivant du mammouth.
L’hybride qui en résulterait, appelé « mammophant », serait adapté à la froide toundra sibérienne et pourrait contribuer à combler le vide écologique laissé par le mammouth lors de son extinction.
Cette technologie présente toutefois des limites et des obstacles qui doivent encore être surmontés. « De nombreux attributs que nous possédons en tant qu’animaux vivants nécessitent plusieurs copies différentes de gènes, mais il n’est pas facile de dire, en regardant un génome reconstruit, combien sont nécessaires », explique Michael Archer, paléontologue à l’université de Nouvelle-Galles du Sud à Sydney, en Australie.
« On croise les doigts pour qu’une seule copie suffise à activer la caractéristique recherchée, mais il y a une grande part d’imprévu dans ces projets. »
La reconstruction du génome n’est toutefois pas la seule méthode que les scientifiques pourraient utiliser pour ressusciter des animaux disparus.
L’auroch, un type de vache préhistorique, est le sujet d’anciennes peintures rupestres dans le monde entier. Il parcourait autrefois les plaines d’Europe et était aussi grand qu’un éléphant.
Il s’est éteint dans les années 1600. Bien qu’il ait disparu depuis longtemps, on trouve encore des gènes d’aurochs dans diverses races de bovins sur le continent, avec des descendants en Espagne, au Portugal, en Italie et dans les Balkans. Les généticiens procèdent actuellement à un « rétrocroisement » de ces espèces afin de produire une descendance plus proche des qualités de l’auroch.
Une autre idée consiste à cloner essentiellement l’animal mort en prélevant le noyau d’une cellule intacte, puis en le transférant dans l’ovule d’un proche parent vivant dans l’espoir qu’un embryon se forme.
Nous l’avons fait des centaines de fois et cela n’a pas marché, puis soudain l’un d’entre eux a marché et nous avons vu cet embryon hybride commencer à se diviser au microscope – Michael Archer
Le problème est qu’il faut une cellule complète pour cela, et que les cellules se décomposent rapidement après la mort. Un animal comme le thylacine, qui s’est éteint il y a près de cent ans, ne pourrait tout simplement pas être ramené à la vie de cette manière.
Mais cela pourrait être une option pour les espèces récemment disparues. En 2003, des chercheurs ont réussi à cloner un bouquetin des Pyrénées, un type de chèvre qui s’est éteint lorsque le dernier individu vivant a été tué par la chute d’un arbre. Malheureusement, le nouveau-né est mort d’une malformation pulmonaire peu après sa naissance.
M. Archer utilise actuellement une variante de la technologie du clonage pour ramener à la vie la grenouille gastro-intestinale du Sud, une espèce originaire du Queensland, qui s’est éteinte en 1983. La créature avait une méthode de reproduction bizarre, avalant ses œufs fécondés et utilisant son estomac comme une sorte d’utérus.
En 2013, il a franchi la première étape : transférer le noyau d’une cellule congelée de grenouille dans l’œuf vide d’un amphibien étroitement apparenté. De façon incroyable, les cellules ont commencé à se diviser, et un embryon s’est formé.
« Nous l’avons fait des centaines de fois sans succès, puis soudain l’une d’entre elles a fonctionné et nous avons vu cet embryon hybride commencer à se diviser sous le microscope, ce qui était très excitant », explique M. Archer.
(…)
Avec BBC






Commentaires Facebook